鸢尾花数据集的探索性数据分析
在本文中,我们将讨论如何对 Iris 数据集进行探索性数据分析。在继续本文之前,我们使用了两个 terns,即 EDA 和 Iris Dataset。让我们看一下有关这些数据集的简介。
什么是探索性数据分析?
探索性数据分析 (EDA)是一种使用一些视觉技术分析数据的技术。使用这种技术,我们可以获得有关数据统计摘要的详细信息。我们还将能够处理重复值、异常值,并查看数据集中存在的一些趋势或模式。
现在让我们简要介绍一下 Iris 数据集。
鸢尾花数据集
如果您具有数据科学背景,那么您都必须熟悉 Iris 数据集。如果你不是那么别担心,我们会在这里讨论这个。
Iris Dataset 被认为是数据科学的 Hello World。它包含五列,即——花瓣长度、花瓣宽度、萼片长度、萼片宽度和物种类型。鸢尾是一种开花植物,研究人员测量了不同鸢尾花的各种特征,并以数字方式记录下来。
注意:这个数据集可以从这里下载。
您可以从上面的链接下载 Iris.csv 文件。现在我们将使用 Pandas 库加载此 CSV 文件,并将其转换为数据帧。 read_csv() 方法用于读取 CSV 文件。
例子:
Python3
import pandas as pd
# Reading the CSV file
df = pd.read_csv("Iris.csv")
# Printing top 5 rows
df.head()Python3
df.shapePython3
df.info()Python3
df.describe()Python3
df.isnull().sum()Python3
data = df.drop_duplicates(subset ="Species",)
dataPython3
df.value_counts("Species")Python3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
sns.countplot(x='Species', data=df, )
plt.show()Python3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
sns.scatterplot(x='SepalLengthCm', y='SepalWidthCm',
hue='Species', data=df, )
# Placing Legend outside the Figure
plt.legend(bbox_to_anchor=(1, 1), loc=2)
plt.show()Python3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
sns.scatterplot(x='PetalLengthCm', y='PetalWidthCm',
hue='Species', data=df, )
# Placing Legend outside the Figure
plt.legend(bbox_to_anchor=(1, 1), loc=2)
plt.show()Python3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
sns.pairplot(df.drop(['Id'], axis = 1),
hue='Species', height=2)Python3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
fig, axes = plt.subplots(2, 2, figsize=(10,10))
axes[0,0].set_title("Sepal Length")
axes[0,0].hist(df['SepalLengthCm'], bins=7)
axes[0,1].set_title("Sepal Width")
axes[0,1].hist(df['SepalWidthCm'], bins=5);
axes[1,0].set_title("Petal Length")
axes[1,0].hist(df['PetalLengthCm'], bins=6);
axes[1,1].set_title("Petal Width")
axes[1,1].hist(df['PetalWidthCm'], bins=6);Python3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
plot = sns.FacetGrid(df, hue="Species")
plot.map(sns.distplot, "SepalLengthCm").add_legend()
plot = sns.FacetGrid(df, hue="Species")
plot.map(sns.distplot, "SepalWidthCm").add_legend()
plot = sns.FacetGrid(df, hue="Species")
plot.map(sns.distplot, "PetalLengthCm").add_legend()
plot = sns.FacetGrid(df, hue="Species")
plot.map(sns.distplot, "PetalWidthCm").add_legend()
plt.show()Python3
data.corr(method='pearson')Python3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
sns.heatmap(df.corr(method='pearson').drop(
['Id'], axis=1).drop(['Id'], axis=0),
annot = True);
plt.show()Python3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
def graph(y):
sns.boxplot(x="Species", y=y, data=df)
plt.figure(figsize=(10,10))
# Adding the subplot at the specified
# grid position
plt.subplot(221)
graph('SepalLengthCm')
plt.subplot(222)
graph('SepalWidthCm')
plt.subplot(223)
graph('PetalLengthCm')
plt.subplot(224)
graph('PetalWidthCm')
plt.show()Python3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
# Load the dataset
df = pd.read_csv('Iris.csv')
sns.boxplot(x='SepalWidthCm', data=df)Python3
# Importing
import sklearn
from sklearn.datasets import load_boston
import pandas as pd
import seaborn as sns
# Load the dataset
df = pd.read_csv('Iris.csv')
# IQR
Q1 = np.percentile(df['SepalWidthCm'], 25,
interpolation = 'midpoint')
Q3 = np.percentile(df['SepalWidthCm'], 75,
interpolation = 'midpoint')
IQR = Q3 - Q1
print("Old Shape: ", df.shape)
# Upper bound
upper = np.where(df['SepalWidthCm'] >= (Q3+1.5*IQR))
# Lower bound
lower = np.where(df['SepalWidthCm'] <= (Q1-1.5*IQR))
# Removing the Outliers
df.drop(upper[0], inplace = True)
df.drop(lower[0], inplace = True)
print("New Shape: ", df.shape)
sns.boxplot(x='SepalWidthCm', data=df)输出:
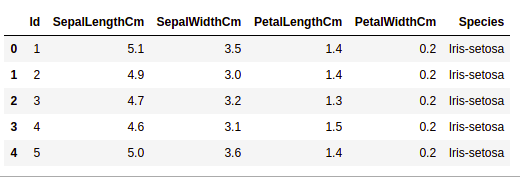
获取有关数据集的信息
我们将使用 shape 参数来获取数据集的形状。
例子:
蟒蛇3
df.shape
输出:
(150, 6)我们可以看到数据框包含 6 列和 150 行。
现在,让我们也了解列及其数据类型。为此,我们将使用 info() 方法。
例子:
蟒蛇3
df.info()
输出:
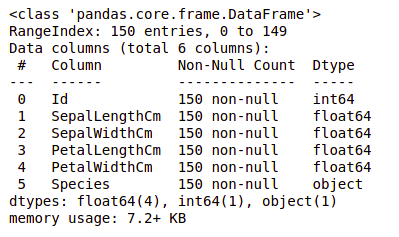
我们可以看到只有一列有分类数据,所有其他列都是数字类型的,没有空条目。
让我们使用describe()方法快速获取数据集的统计摘要。 describe()函数对数据集应用基本的统计计算,如极值、数据点标准偏差计数等。任何缺失值或 NaN 值都会自动跳过。 describe()函数可以很好地描述数据的分布。
例子:
蟒蛇3
df.describe()
输出:
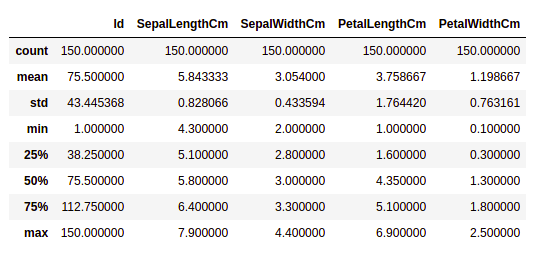
我们可以看到每列的计数以及它们的平均值、标准偏差、最小值和最大值。
检查缺失值
我们将检查我们的数据是否包含任何缺失值。当没有为一个或多个项目或整个单元提供信息时,可能会出现缺失值。我们将使用 isnull() 方法。
例子:
蟒蛇3
df.isnull().sum()
输出:
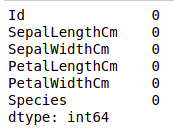
我们可以看到没有列作为任何缺失值。
注意:有关更多信息,请参阅在 Pandas 中处理缺失数据。
检查重复项
让我们看看我们的数据集是否包含任何重复项。 Pandas drop_duplicates() 方法有助于从数据框中删除重复项。
例子:
蟒蛇3
data = df.drop_duplicates(subset ="Species",)
data
输出:
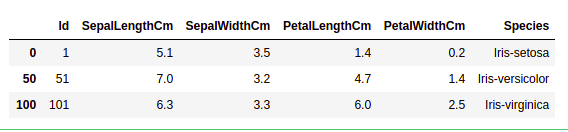
我们可以看到只有三个独特的物种。让我们看看数据集是否平衡,即所有物种是否包含相等数量的行。我们将使用 Series.value_counts()函数。此函数返回一个包含唯一值计数的系列。
例子:
蟒蛇3
df.value_counts("Species")
输出:
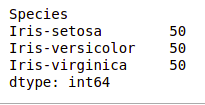
我们可以看到所有物种都包含相等数量的行,因此我们不应删除任何条目。
数据可视化
可视化目标列
我们的目标列将是 Species 列,因为最后我们将只需要根据物种的结果。让我们看看物种的计数图。
Note: We will use Matplotlib and Seaborn library for the data visulalization. If you want to know about these modules refer to the articles –
- Matplotlib Tutorial
- Python Seaborn Tutorial
例子:
蟒蛇3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
sns.countplot(x='Species', data=df, )
plt.show()
输出:
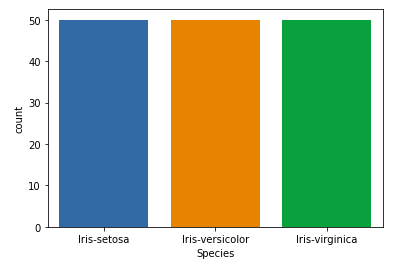
变量之间的关系
我们将看到萼片长度和萼片宽度之间的关系以及花瓣长度和花瓣宽度之间的关系。
示例 1:比较萼片长度和萼片宽度
蟒蛇3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
sns.scatterplot(x='SepalLengthCm', y='SepalWidthCm',
hue='Species', data=df, )
# Placing Legend outside the Figure
plt.legend(bbox_to_anchor=(1, 1), loc=2)
plt.show()
输出:
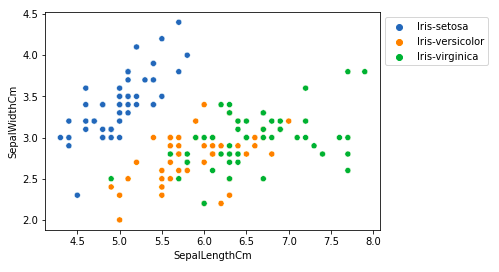
从上图,我们可以推断——
- 种 Setosa 具有较小的萼片长度但较大的萼片宽度。
- Versicolor Species 在萼片长度和宽度方面位于其他两个物种的中间
- 种 Virginica 具有较大的萼片长度但较小的萼片宽度。
示例 2:比较花瓣长度和花瓣宽度
蟒蛇3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
sns.scatterplot(x='PetalLengthCm', y='PetalWidthCm',
hue='Species', data=df, )
# Placing Legend outside the Figure
plt.legend(bbox_to_anchor=(1, 1), loc=2)
plt.show()
输出:
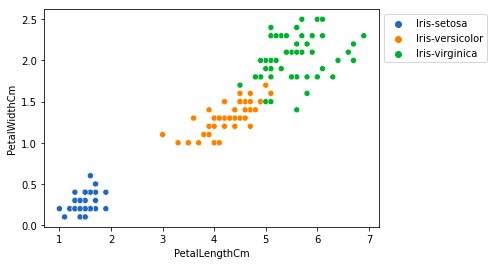
从上图,我们可以推断——
- 种 Setosa 具有较小的花瓣长度和宽度。
- Versicolor Species 在花瓣长度和宽度方面位于其他两个物种的中间
- 种类 Virginica 具有最大的花瓣长度和宽度。
让我们使用配对图绘制所有列的关系。它可用于多变量分析。
例子:
蟒蛇3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
sns.pairplot(df.drop(['Id'], axis = 1),
hue='Species', height=2)
输出:
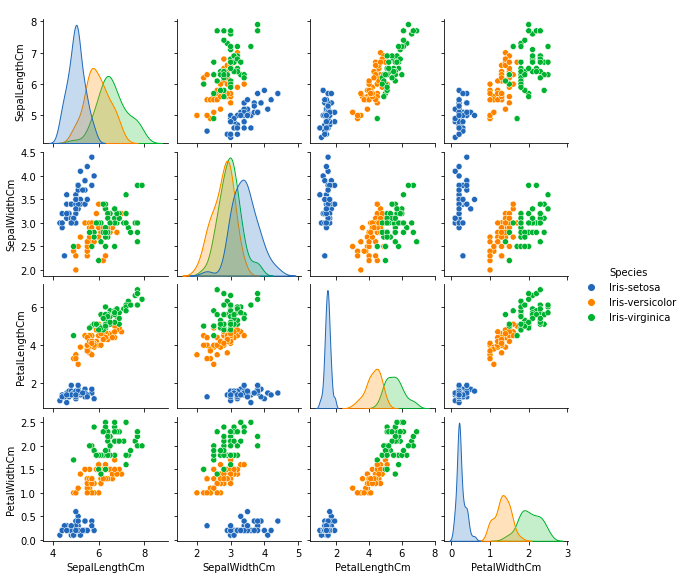
我们可以从这个图中看到许多类型的关系,例如 Seotsa 物种的花瓣宽度和长度最小。它也具有最小的萼片长度但较大的萼片宽度。可以收集有关任何其他物种的此类信息。
直方图
直方图允许查看各个列的数据分布。它可用于单变量和双变量分析。
例子:
蟒蛇3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
fig, axes = plt.subplots(2, 2, figsize=(10,10))
axes[0,0].set_title("Sepal Length")
axes[0,0].hist(df['SepalLengthCm'], bins=7)
axes[0,1].set_title("Sepal Width")
axes[0,1].hist(df['SepalWidthCm'], bins=5);
axes[1,0].set_title("Petal Length")
axes[1,0].hist(df['PetalLengthCm'], bins=6);
axes[1,1].set_title("Petal Width")
axes[1,1].hist(df['PetalWidthCm'], bins=6);
输出:
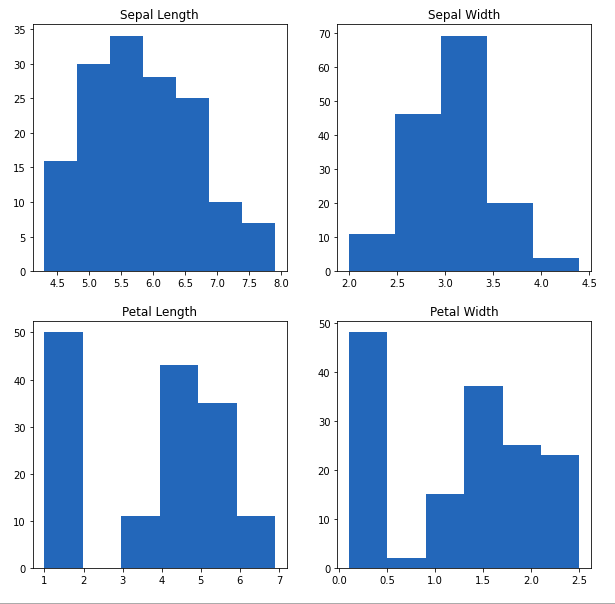
从上图我们可以看出——
- 萼片长度的最高频率在 30 到 35 之间,在 5.5 到 6 之间
- 萼片宽度的最高频率约为 70,介于 3.0 和 3.5 之间
- 花瓣长度的最高频率在 50 左右,介于 1 和 2 之间
- 花瓣宽度的最高频率在40到50之间,在0.0到0.5之间
带有 Distplot 图的直方图
Distplot 基本上用于观察的单变量集,并通过直方图对其进行可视化,即只有一个观察,因此我们选择数据集的一个特定列。
例子:
蟒蛇3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
plot = sns.FacetGrid(df, hue="Species")
plot.map(sns.distplot, "SepalLengthCm").add_legend()
plot = sns.FacetGrid(df, hue="Species")
plot.map(sns.distplot, "SepalWidthCm").add_legend()
plot = sns.FacetGrid(df, hue="Species")
plot.map(sns.distplot, "PetalLengthCm").add_legend()
plot = sns.FacetGrid(df, hue="Species")
plot.map(sns.distplot, "PetalWidthCm").add_legend()
plt.show()
输出:
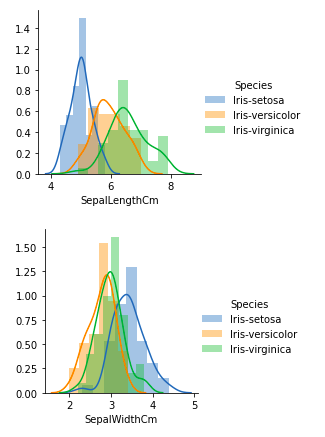
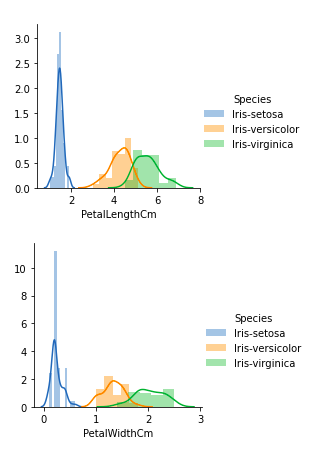
从上面的图中,我们可以看到——
- 在萼片长度的情况下,有大量的重叠。
- 在萼片宽度的情况下,也有大量的重叠。
- 在花瓣长度的情况下,重叠量很少。
- 在花瓣宽度的情况下,重叠也很少。
所以我们可以使用花瓣长度和花瓣宽度作为分类特征。
处理相关性
Pandas dataframe.corr() 用于查找数据帧中所有列的成对相关性。任何 NA 值都会被自动排除。对于数据框中的任何非数字数据类型列,它都会被忽略。
例子:
蟒蛇3
data.corr(method='pearson')
输出:
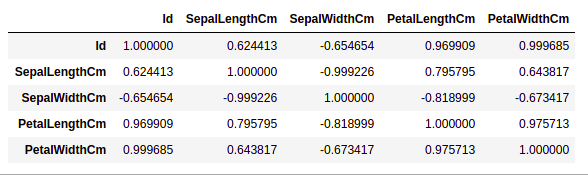
热图
热图是一种数据可视化技术,用于将数据集分析为二维颜色。基本上,它显示了数据集中所有数值变量之间的相关性。简单来说,我们可以使用热图绘制上述相关性。
例子:
蟒蛇3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
sns.heatmap(df.corr(method='pearson').drop(
['Id'], axis=1).drop(['Id'], axis=0),
annot = True);
plt.show()
输出:
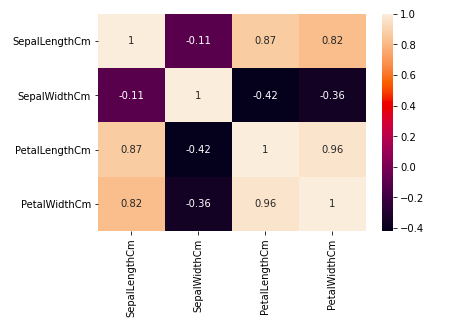
从上图我们可以看出——
- 花瓣宽度和花瓣长度具有高度相关性。
- 花瓣长度和萼片宽度有很好的相关性。
- 花瓣宽度和萼片长度具有良好的相关性。
箱线图
我们可以使用箱线图来查看分类值 os 如何与其他数值分布。
例子:
蟒蛇3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
def graph(y):
sns.boxplot(x="Species", y=y, data=df)
plt.figure(figsize=(10,10))
# Adding the subplot at the specified
# grid position
plt.subplot(221)
graph('SepalLengthCm')
plt.subplot(222)
graph('SepalWidthCm')
plt.subplot(223)
graph('PetalLengthCm')
plt.subplot(224)
graph('PetalWidthCm')
plt.show()
输出:
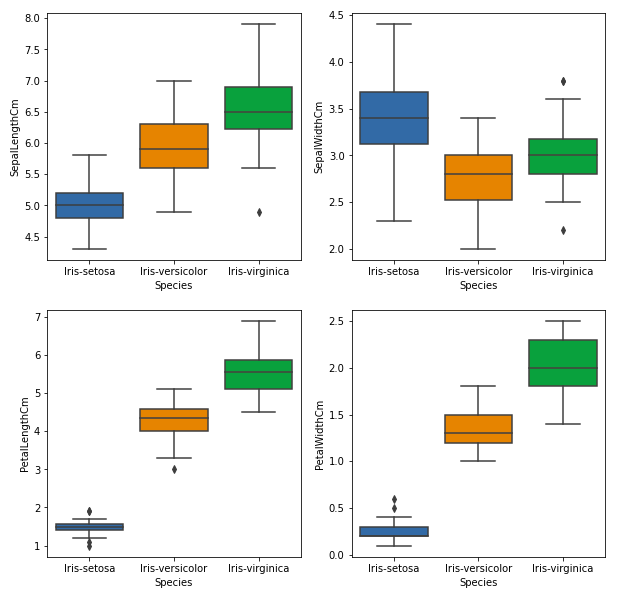
从上图我们可以看出——
- Species Setosa 的特征最小,分布较少,有一些异常值。
- 物种 Versicolor 具有平均特征。
- 物种 Virginica 具有最高的特征
处理异常值
异常值是与其余(所谓的正常)对象显着不同的数据项/对象。它们可能是由测量或执行错误引起的。异常值检测的分析称为异常值挖掘。检测异常值的方法有很多种,去除过程是数据帧,与从熊猫的数据帧中去除数据项相同。
让我们考虑 iris 数据集,并绘制 SepalWidthCm 列的箱线图。
例子:
蟒蛇3
# importing packages
import seaborn as sns
import matplotlib.pyplot as plt
# Load the dataset
df = pd.read_csv('Iris.csv')
sns.boxplot(x='SepalWidthCm', data=df)
输出:
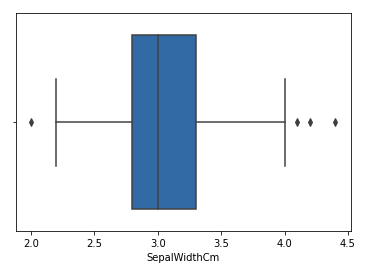
在上图中,大于 4 和小于 2 的值充当异常值。
去除异常值
为了去除异常值,必须遵循使用其在数据集中的确切位置从数据集中删除条目的相同过程,因为在上述所有检测异常值的方法中,最终结果是满足异常值定义的所有数据项的列表根据使用的方法。
示例:我们将使用 IQR 检测异常值,然后将其删除。我们还将绘制箱线图以查看是否删除了异常值。
蟒蛇3
# Importing
import sklearn
from sklearn.datasets import load_boston
import pandas as pd
import seaborn as sns
# Load the dataset
df = pd.read_csv('Iris.csv')
# IQR
Q1 = np.percentile(df['SepalWidthCm'], 25,
interpolation = 'midpoint')
Q3 = np.percentile(df['SepalWidthCm'], 75,
interpolation = 'midpoint')
IQR = Q3 - Q1
print("Old Shape: ", df.shape)
# Upper bound
upper = np.where(df['SepalWidthCm'] >= (Q3+1.5*IQR))
# Lower bound
lower = np.where(df['SepalWidthCm'] <= (Q1-1.5*IQR))
# Removing the Outliers
df.drop(upper[0], inplace = True)
df.drop(lower[0], inplace = True)
print("New Shape: ", df.shape)
sns.boxplot(x='SepalWidthCm', data=df)
输出:
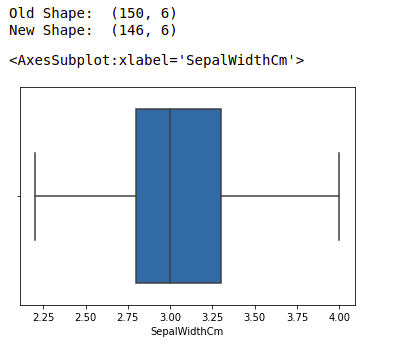
注意:有关更多信息,请参阅使用Python检测和删除异常值