使用Python材料分析
使用一个以上领域的基本原理来解决一个复杂的问题可能很难通过使用一个领域的知识来达到。通过这种方法,人们还可以重新定义通常边界之外的问题,并使用对早期认为不可能获得的难以理解的情况的新理解来找到解决方案。
材料分析
通俗地说,分析材料及其特性是一个研究领域。
科学 - 这是一项深入了解材料基本特性的研究,以确定该材料是否适合其预期用例或需要一些掺杂(或任何其他方法)以使其完全符合目的。
用例:这项研究还与计算机科学相结合,以在没有太多实际实施的情况下获得更好、更精确的数据洞察。
示例:如果有一个数据库,例如,Mn(锰)化合物及其磁性行为。分析相同的数据可以使用机器学习方法预测未知磁性化合物(其特性仍然是新的)的磁性。
描述符
在字典中,它被描述为用于描述或识别某物的词或表达。
描述符用于描述计算算法的化合物。许多属性元素的表示可以转换为向量和矩阵的数学格式(例如使用单热向量编码来描述元素的电子配置),以将它们作为输入传递给机器学习算法。
Pymatgen 模块
Pymatgen 是Python材料基因组学的缩写形式。它是一个强大的、开源的、广泛使用的用于材料分析的Python库。
注意 - 仅获取电子配置、原子序数或任何其他非常基本的材料属性并不考虑材料分析。
Pymatgen 受到广泛青睐,因为它是:
- 用于表示元素、站点、分子、结构对象、最近邻的高度灵活的类。
- 各种输入/输出格式,如 CIF、Gaussian、XYZ、VASP。
- 电子结构分析,例如态密度和能带结构。
- 强大的分析工具。
- 与 Materials Project REST API、晶体学开放数据库和其他外部数据源集成。
- 它可以免费使用、有据可查、开放且快速。
安装
由于它不是内置的Python库,所以需要在外部安装它。
第一种方法:
最直接的安装是使用 conda。安装 conda 后:
conda install –channel conda-forge pymatgen
Pymatgen 使用 'gcc' 进行编译,因此编译 pymatgen 需要相同的最新版本。
conda install gcc
Pymatgen 是开源的,因此会定期添加新功能。因此,要将 pymatgen 升级到最新版本:
conda upgrade pymatgen
第二种方法:
使用点子:
pip install pymatgen
并升级pymatgen
pip install –upgrade pymatgen
第三种方法:
在 google collab 上安装 pymatgen
!pip install pymatgen
执行
元素和化合物的详细信息
使用 Pymatgen 库的 Element 类获取元素的详细信息(如原子质量、熔点)。将元素符号作为参数传递给 Element 类。
同样,也可以得到一个化合物的详细信息。
Python3
import pymatgen.core as pg
# Fetch details of an Element
fe = pg.Element("Fe")
# Atomic mass
print('atomic mass: ', fe.atomic_mass)
print('atomic mass: ', fe.Z)
# Melting point
print('melting poin: ', fe.melting_point)
# Fetch details of a composition
cmps = pg.Composition("NaCl")
print('weight of composition: ', cmps.weight)
# Composition allows strings to
# be treated as an Element object
# It returns the number of Cl
# atoms present in the composition
cmps["Cl"]Python3
# import module
import pymatgen.core as pg
from pymatgen.symmetry.analyzer import SpacegroupAnalyzer
# assign and display data
lattice = pg.Lattice.cubic(4.2)
print('LATTICE\n', lattice, '\n')
structure = pg.Structure(lattice, ["Li", "Cl"],
[[0, 0, 0],
[0.5, 0.5, 0.5]])
print('STRUCTURE', '\n', structure)
# Convert structure of the compound
# to user defined formats
structure.to(fmt="poscar")
structure.to(filename="POSCAR")
structure.to(filename="CsCl.cif")Python3
# Reading a structure from a file
structure = pg.Structure.from_str(open("MnO2.cif").read(),
fmt="cif")
structure = pg.Structure.from_file("MnO2.cif")
# Reading a molecule from a file
graphite = pg.Molecule.from_file("graphite.xyz")
# Writing the same molecule but in other file format
mol.to("graphite.cif")Python3
# import module
from pymatgen.ext.matproj import MPRester
# create object
m = MPRester(API_key)
# fetch all the required properties of an element using mpid
# fetching details of a compound related to TaskId=mpid-1010
data_one = m.query(criteria={'task_id': 'mp-1010'},
properties=["full_formula",
"spacegroup.symbol",
"volume",
"nsites", "density",
"spacegroup.crystal_system",
"final_energy_per_atom",
"final_energy"])
# display fetched data
print(data_one)Python3
# Fetch all the compounds details of an element in the database
# Fetching data of Fe-Iron
from pymatgen.ext.matproj import MPRester
import pandas as pd
m = MPRester(API_key)
data_s = m.query(criteria={"elements": {"$in": ["Fe"]}},
properties=["full_formula",
"spacegroup.symbol",
"volume",
"nsites", "density",
"spacegroup.crystal_system",
"final_energy_per_atom",
"final_energy"])
# convert data to pandas data
# frame and store it in .csv file
df = pd.DataFrame(data_s)
df.to_csv('all.csv')
# display data saved in all.csv
print(df)Python3
# import module
from pymatgen.core import Structure
from pymatgen.ext.matproj import MPRester
import re
m = MPRester(API_key)
id = 'mp-1183751'
data_c = m.query(criteria={'task_id': id},
properties=[
'cifs.conventional_standard'])
# delete extras
with open('cnt.cif', 'w') as f:
filedata = str(data_c)
filedata = re.sub(r'.*_occupancy',
'', filedata)
filedata = filedata[:-4]
filedata = filedata.replace('\\n',
'\n')
f.write(filedata[1:])
f.close()
count = len(open('cnt.cif',
'r').readlines())
# display the no. of atoms
print(count)输出:

结构和文件格式
Pymatgen 有许多库,它们根据它们所代表的属性进行分组/分离。在这里,创建 pymatgen 第一个对角点阵矩阵,然后获取其结构。如果没有文件名,则返回一个字符串。否则,输出将写入文件。如果只提供文件名
蟒蛇3
# import module
import pymatgen.core as pg
from pymatgen.symmetry.analyzer import SpacegroupAnalyzer
# assign and display data
lattice = pg.Lattice.cubic(4.2)
print('LATTICE\n', lattice, '\n')
structure = pg.Structure(lattice, ["Li", "Cl"],
[[0, 0, 0],
[0.5, 0.5, 0.5]])
print('STRUCTURE', '\n', structure)
# Convert structure of the compound
# to user defined formats
structure.to(fmt="poscar")
structure.to(filename="POSCAR")
structure.to(filename="CsCl.cif")
输出:
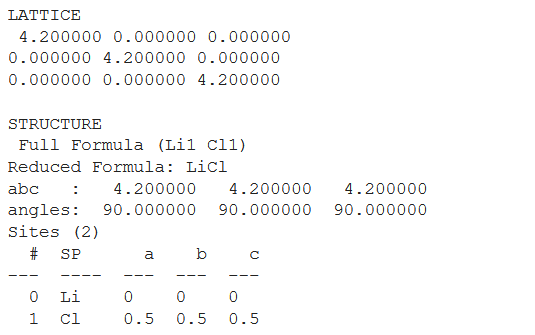
获取结构
Pymatgen 还允许用户从外部文件中读取结构。可以使用以下代码中使用的字符串和文件以两种方式实现相同的目的。我们要获取的文件是 MnO2.cif 的计算版本。
蟒蛇3
# Reading a structure from a file
structure = pg.Structure.from_str(open("MnO2.cif").read(),
fmt="cif")
structure = pg.Structure.from_file("MnO2.cif")
# Reading a molecule from a file
graphite = pg.Molecule.from_file("graphite.xyz")
# Writing the same molecule but in other file format
mol.to("graphite.cif")
输出:

它还可以用作文件转换器,因为它允许从一种格式的文件中读取分子并将相同的分子写入另一种格式的文件中。
外部数据源
如上所述,pymatgen 可以链接到不同的外部数据源。可以使用项目的 MPRester API 在 pymatgen 中访问 Material 项目的数据。
材料项目是通过开放的材料应用程序编程接口 API(也称为 MPRester API,因为它基于 REpresentational State Transfer (REST) 原则)提供其数据和科学分析的外部数据库之一。可以想象,该 API 可以与支持基本 HTTP 请求的任何编程语言一起使用,已在 pymatgen 库中实现了 MPRester API 的包装器,以方便想要利用其数据的研究人员。
API 密钥生成请参考此网站 -> https://materialsproject.org/open
首先,对象是由 API 密钥创建的,然后查询特定任务 id 的属性数据(任务 id 可以被认为是 Material Project 数据库中存在的每个元素的唯一标识)。
注意 - 属性名称在属性下提到。如果不存在这样的特定属性数据,则针对该特定属性接收空对象。
蟒蛇3
# import module
from pymatgen.ext.matproj import MPRester
# create object
m = MPRester(API_key)
# fetch all the required properties of an element using mpid
# fetching details of a compound related to TaskId=mpid-1010
data_one = m.query(criteria={'task_id': 'mp-1010'},
properties=["full_formula",
"spacegroup.symbol",
"volume",
"nsites", "density",
"spacegroup.crystal_system",
"final_energy_per_atom",
"final_energy"])
# display fetched data
print(data_one)
输出格式在字典数据结构中,便于访问所需的属性
输出:


其次,获取铁(Fe)化合物的所有定义的属性数据(元素和化合物)。
蟒蛇3
# Fetch all the compounds details of an element in the database
# Fetching data of Fe-Iron
from pymatgen.ext.matproj import MPRester
import pandas as pd
m = MPRester(API_key)
data_s = m.query(criteria={"elements": {"$in": ["Fe"]}},
properties=["full_formula",
"spacegroup.symbol",
"volume",
"nsites", "density",
"spacegroup.crystal_system",
"final_energy_per_atom",
"final_energy"])
# convert data to pandas data
# frame and store it in .csv file
df = pd.DataFrame(data_s)
df.to_csv('all.csv')
# display data saved in all.csv
print(df)
查询Fe元素所有数据的输出是嵌套字典格式,并且非常大,无法显示在控制台中,因此首先将其转换为pandas数据框,然后将其保存为.csv文件。
输出:


现实生活用例
在这里,我们将计算化合物中的原子数。它可以通过以 CIF 格式获取化合物的结构细节来轻松完成,因为它包含化合物的每个原子的所有坐标位置。
首先,从文件中删除所有不必要的文本,然后计算剩余的行数。
CoNi3 化合物与 mp-1183751 相关
蟒蛇3
# import module
from pymatgen.core import Structure
from pymatgen.ext.matproj import MPRester
import re
m = MPRester(API_key)
id = 'mp-1183751'
data_c = m.query(criteria={'task_id': id},
properties=[
'cifs.conventional_standard'])
# delete extras
with open('cnt.cif', 'w') as f:
filedata = str(data_c)
filedata = re.sub(r'.*_occupancy',
'', filedata)
filedata = filedata[:-4]
filedata = filedata.replace('\\n',
'\n')
f.write(filedata[1:])
f.close()
count = len(open('cnt.cif',
'r').readlines())
# display the no. of atoms
print(count)
输出:
4