R 编程中的有监督和无监督学习
人工智能和计算机游戏领域的先驱Arthur Samuel创造了“机器学习”一词。他将机器学习定义为—— “使计算机能够在没有明确编程的情况下学习的研究领域” 。以一种非常外行的方式,机器学习(ML) 可以解释为根据计算机的经验自动化和改进计算机的学习过程,而无需实际编程,即没有任何人工帮助。该过程从提供高质量数据开始,然后通过使用数据和不同算法构建机器学习模型来训练我们的机器(计算机)。算法的选择取决于我们拥有什么类型的数据以及我们试图自动化的任务类型。主要有四种学习方式。

在本文中,让我们讨论两个最重要的学习,例如 R 编程中的监督学习和无监督学习。
R 语言基本上是由统计学家开发的,旨在帮助其他统计学家和开发人员更快、更高效地处理数据。到目前为止,我们知道机器学习基本上是处理大量数据和统计数据,作为数据科学的一部分,始终建议使用 R 语言。因此,R 语言对于那些使用机器学习使任务更容易、更快和创新的人来说越来越方便。以下是 R 语言在 R 编程中实现机器学习算法的一些主要优势。
使用 R 语言实现机器学习的优势
- 它提供了很好的解释性代码。例如,如果您正处于机器学习项目的早期工作,并且需要解释您所做的工作,那么将 R 语言与Python语言进行比较就变得很容易,因为它提供了处理数据的正确统计方法用更少的代码行。
- R语言非常适合数据可视化。 R 语言提供了使用机器学习模型的最佳原型。
- R 语言拥有最好的工具和库包来处理机器学习项目。开发人员可以使用这些包来创建机器学习项目的最佳前模型、模型和后模型。此外,R 的包比Python语言更先进和更广泛,这使其成为处理机器学习项目的首选。
监督学习
顾名思义,监督学习表明存在监督者作为教师。基本上,监督学习是一种学习,在这种学习中,我们使用标记良好的数据来教授或训练机器,这意味着某些数据已经用正确答案标记了。之后,机器被提供一组新的示例(数据),以便监督学习算法分析训练数据(训练示例集)并从标记数据中产生正确的结果。监督学习分为两类算法:
- 分类:分类问题是当输出变量是一个类别时,例如“红色”或“蓝色”或“疾病”和“无疾病”。
- 回归:回归问题是当输出变量是真实值时,例如“美元”或“重量”。
监督学习处理或学习“标记”数据。这意味着某些数据已经被标记为正确答案。
类型
- 回归
- 逻辑回归
- 分类
- 朴素贝叶斯分类器
- 决策树
- 支持向量机
在 R 中的实现
让我们在 R 编程中实现一种非常流行的监督学习,即简单线性回归。简单线性回归是一种统计方法,它使我们能够总结和研究两个连续(定量)变量之间的关系。一个用 x 表示的变量被认为是一个自变量,另一个用 y 表示的变量被认为是一个因变量。假设两个变量是线性相关的。因此,我们试图找到一个线性函数,它尽可能准确地预测响应值(y)作为特征或自变量(x)的函数。 R 中回归分析的基本语法是:
Syntax:
lm(Y ~ model)
where,
Y is the object containing the dependent variable to be predicted and model is the formula for the chosen mathematical model.
命令lm()提供模型的系数,但没有进一步的统计信息。以下 R 代码用于实现简单的线性回归。
例子:
R
# Simple Linear Regression
# Importing the dataset
dataset = read.csv('salary.csv')
# Splitting the dataset into the
# Training set and Test set
install.packages('caTools')
library(caTools)
split = sample.split(dataset$Salary,
SplitRatio = 0.7)
trainingset = subset(dataset, split == TRUE)
testset = subset(dataset, split == FALSE)
# Fitting Simple Linear Regression
# to the Training set
lm.r = lm(formula = Salary ~ YearsExperience,
data = trainingset)
coef(lm.r)
# Predicting the Test set results
ypred = predict(lm.r, newdata = testset)
install.packages("ggplot2")
library(ggplot2)
# Visualising the Training set results
ggplot() + geom_point(aes(x = trainingset$YearsExperience,
y = trainingset$Salary),
colour = 'red') +
geom_line(aes(x = trainingset$YearsExperience,
y = predict(lm.r, newdata = trainingset)),
colour = 'blue') +
ggtitle('Salary vs Experience (Training set)') +
xlab('Years of experience') +
ylab('Salary')
# Visualising the Test set results
ggplot() + geom_point(aes(x = testset$YearsExperience,
y = testset$Salary),
colour = 'red') +
geom_line(aes(x = trainingset$YearsExperience,
y = predict(lm.r, newdata = trainingset)),
colour = 'blue') +
ggtitle('Salary vs Experience (Test set)') +
xlab('Years of experience') +
ylab('Salary')R
# Installing Packages
install.packages("ClusterR")
install.packages("cluster")
# Loading package
library(ClusterR)
library(cluster)
# Loading data
data(iris)
# Structure
str(iris)
# Removing initial label of
# Species from original dataset
iris_1 <- iris[, -5]
# Fitting K-Means clustering Model
# to training dataset
set.seed(240) # Setting seed
kmeans.re <- kmeans(iris_1, centers = 3,
nstart = 20)
kmeans.re
# Cluster identification for
# each observation
kmeans.re$cluster
# Confusion Matrix
cm <- table(iris$Species, kmeans.re$cluster)
cm
# Model Evaluation and visualization
plot(iris_1[c("Sepal.Length", "Sepal.Width")])
plot(iris_1[c("Sepal.Length", "Sepal.Width")],
col = kmeans.re$cluster)
plot(iris_1[c("Sepal.Length", "Sepal.Width")],
col = kmeans.re$cluster,
main = "K-means with 3 clusters")
# Plotiing cluster centers
kmeans.re$centers
kmeans.re$centers[, c("Sepal.Length",
"Sepal.Width")]
# cex is font size, pch is symbol
points(kmeans.re$centers[, c("Sepal.Length",
"Sepal.Width")],
col = 1:3, pch = 8, cex = 3)
# Visualizing clusters
y_kmeans <- kmeans.re$cluster
clusplot(iris_1[, c("Sepal.Length", "Sepal.Width")],
y_kmeans,
lines = 0,
shade = TRUE,
color = TRUE,
labels = 2,
plotchar = FALSE,
span = TRUE,
main = paste("Cluster iris"),
xlab = 'Sepal.Length',
ylab = 'Sepal.Width')输出:
Intercept YearsExperience
24558.39 10639.23
可视化训练集结果:
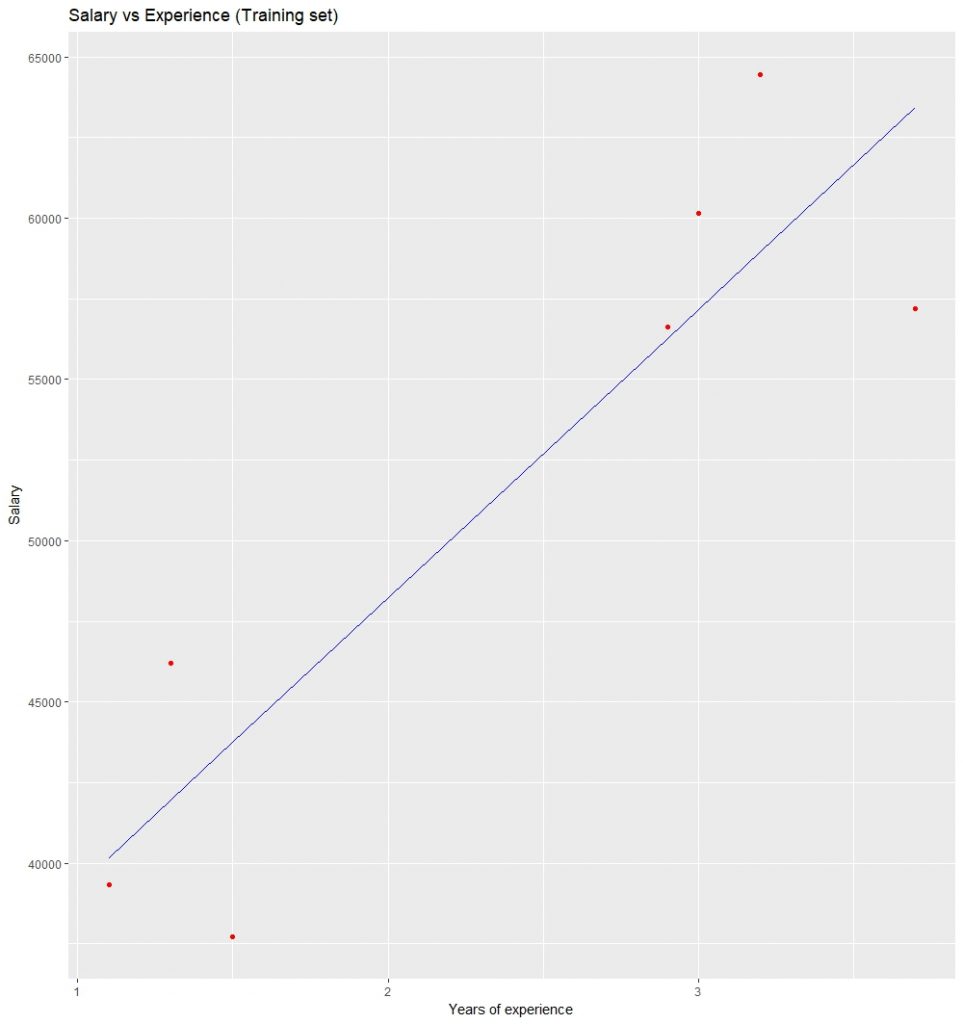
可视化测试集结果:
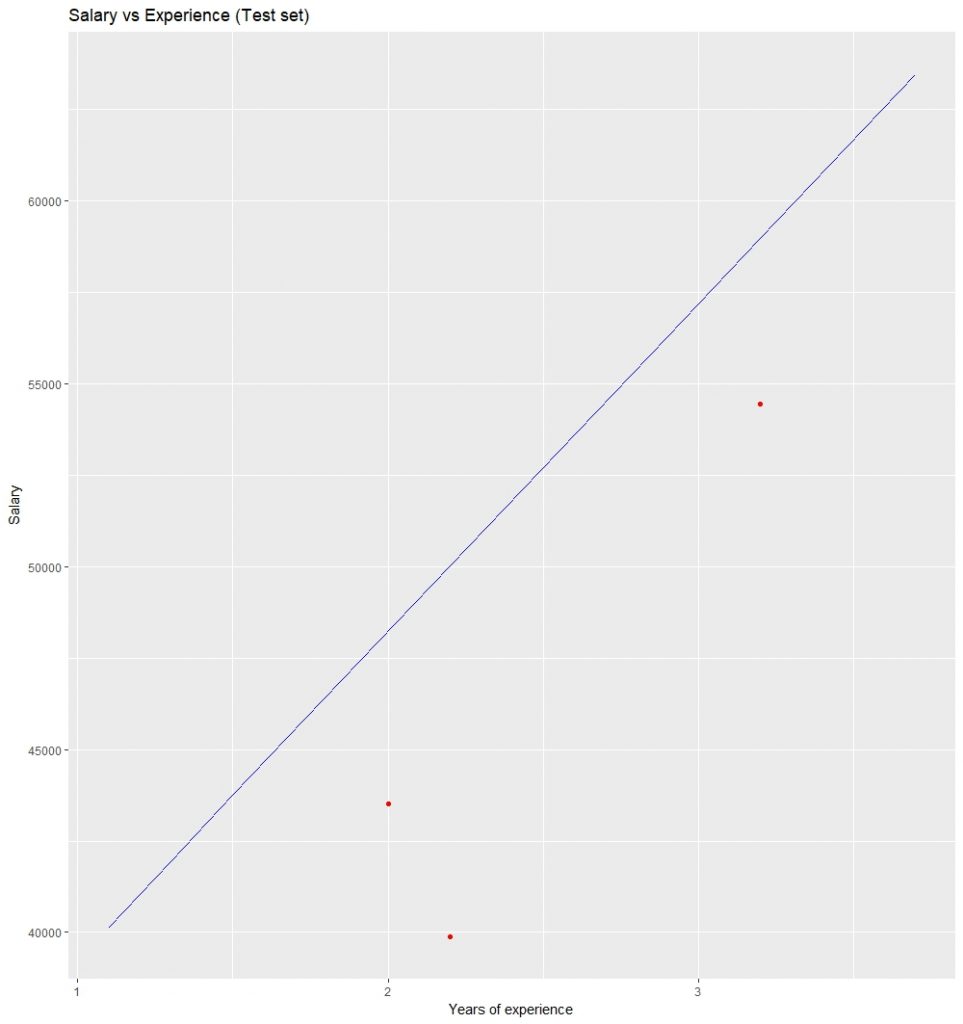
无监督学习
无监督学习是使用既未分类也未标记的信息训练机器,并允许算法在没有指导的情况下对该信息采取行动。在这里,机器的任务是根据相似性、模式和差异对未分类的信息进行分组,而无需事先对数据进行任何训练。与监督学习不同,不提供任何老师,这意味着不会对机器进行任何培训。因此,机器被限制在我们自己寻找未标记数据中的隐藏结构。无监督学习分为两类算法:
- 聚类:聚类问题是您想要发现数据中固有的分组,例如按购买行为对客户进行分组。
- 关联:关联规则学习问题是您想要发现描述大部分数据的规则,例如购买 X 的人也倾向于购买 Y。
类型
聚类:
- 独占(分区)
- 凝聚的
- 重叠
- 概率的
聚类类型:
- 层次聚类
- K均值聚类
- K-NN(k 个最近邻)
- 主成分分析
- 奇异值分解
- 独立成分分析
在 R 中的实现
让我们在 R 编程中实现一种非常流行的无监督学习,即 K 均值聚类。 R 编程中的 K 表示聚类是一种无监督非线性算法,它基于相似性或相似组对数据进行聚类。它试图将观察结果划分为预先指定数量的集群。发生数据分割以将每个训练示例分配给称为集群的段。在无监督算法中,给出了对原始数据的高度依赖,并且给出了大量的人工审查以进行相关性审查。它用于银行、医疗保健、零售、媒体等各个领域。
例子:
电阻
# Installing Packages
install.packages("ClusterR")
install.packages("cluster")
# Loading package
library(ClusterR)
library(cluster)
# Loading data
data(iris)
# Structure
str(iris)
# Removing initial label of
# Species from original dataset
iris_1 <- iris[, -5]
# Fitting K-Means clustering Model
# to training dataset
set.seed(240) # Setting seed
kmeans.re <- kmeans(iris_1, centers = 3,
nstart = 20)
kmeans.re
# Cluster identification for
# each observation
kmeans.re$cluster
# Confusion Matrix
cm <- table(iris$Species, kmeans.re$cluster)
cm
# Model Evaluation and visualization
plot(iris_1[c("Sepal.Length", "Sepal.Width")])
plot(iris_1[c("Sepal.Length", "Sepal.Width")],
col = kmeans.re$cluster)
plot(iris_1[c("Sepal.Length", "Sepal.Width")],
col = kmeans.re$cluster,
main = "K-means with 3 clusters")
# Plotiing cluster centers
kmeans.re$centers
kmeans.re$centers[, c("Sepal.Length",
"Sepal.Width")]
# cex is font size, pch is symbol
points(kmeans.re$centers[, c("Sepal.Length",
"Sepal.Width")],
col = 1:3, pch = 8, cex = 3)
# Visualizing clusters
y_kmeans <- kmeans.re$cluster
clusplot(iris_1[, c("Sepal.Length", "Sepal.Width")],
y_kmeans,
lines = 0,
shade = TRUE,
color = TRUE,
labels = 2,
plotchar = FALSE,
span = TRUE,
main = paste("Cluster iris"),
xlab = 'Sepal.Length',
ylab = 'Sepal.Width')
输出:
- 模型 kmeans_re:
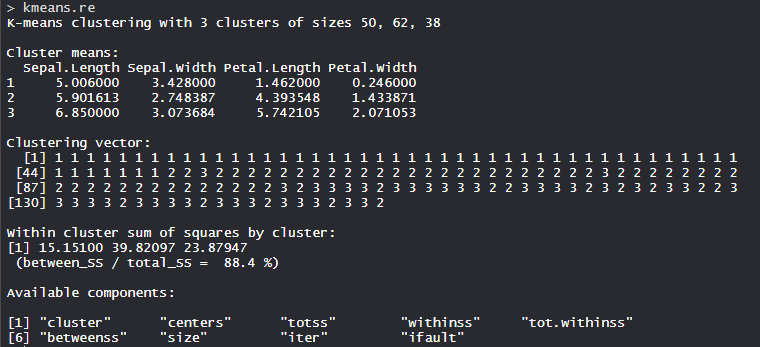
制作了 3 个簇,分别具有 50、62 和 38 种尺寸。在集群内,平方和为 88.4%。
- 集群识别:
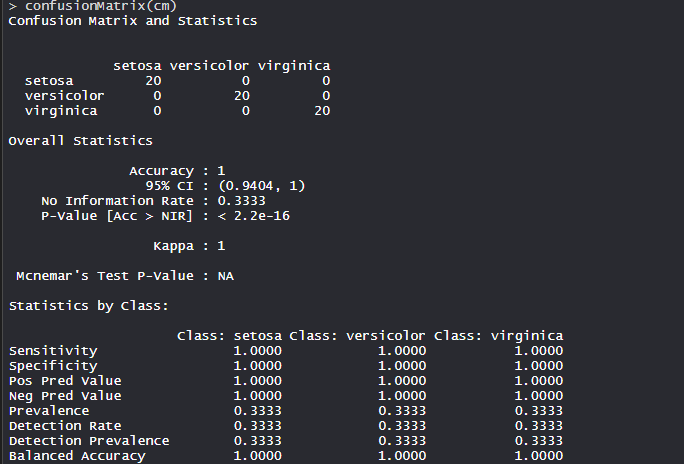
该模型达到了 100% 的准确度,p 值小于 1。这表明该模型是好的。
- 混淆矩阵:
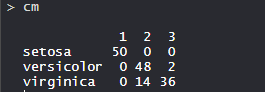
因此,50 Setosa 被正确分类为 Setosa。在 62 种 Versicolor 中,48 种 Versicolor 被正确归类为 Versicolor,14 种被归类为 virginica。在 36 个 virginica 中,19 个 virginica 被正确归类为 virginica,2 个被归类为 Versicolor。
- 具有 3 个聚类图的 K 均值:
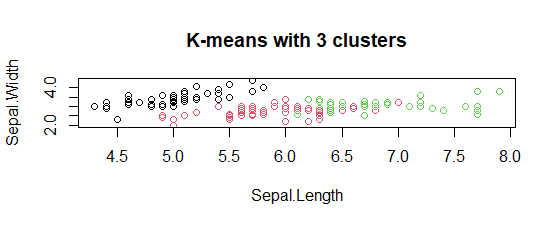
该模型显示了 3 个具有三种不同颜色、Sepal.length 和 Sepal.width 的聚类图。
- 绘制聚类中心:
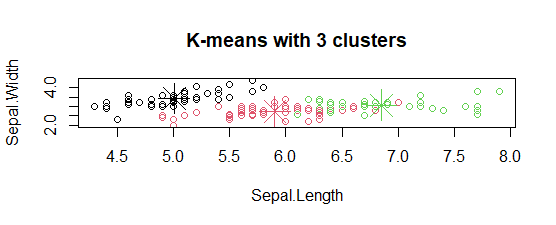
在图中,簇的中心用与簇颜色相同的十字符号标记。
- 集群图:
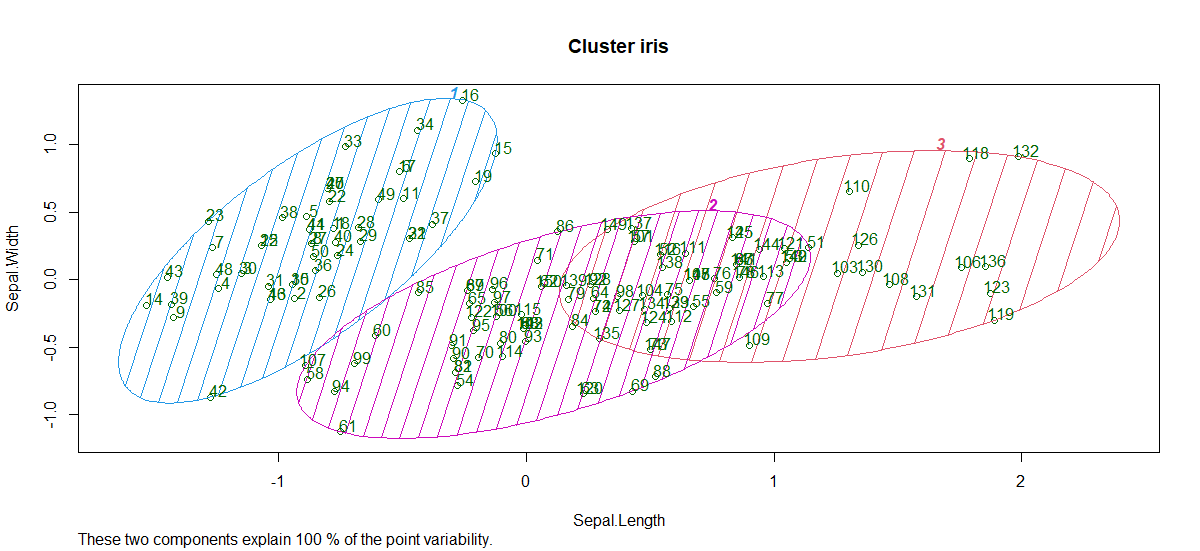
因此,形成了具有不同萼片长度和萼片宽度的 3 个簇。