R中的生存分析
R 编程语言中的生存分析处理特定时间事件的预测。它处理在指定时间内发生的有趣事件,并且它的失败会产生审查观察,即不完整的观察。
R 编程语言中的生存分析
生物科学是生存分析最重要的应用,我们可以在其中预测生物的时间,例如。当它们乘以大小等时。
用于进行生存分析的方法:
有两种方法可用于在 R 编程语言中执行生存分析:
- Kaplan-Meier 方法
- Cox 比例风险模型
卡普兰-迈耶法
Kaplan-Meier 方法用于使用 Kaplan-Meier 估计器对截断或删失数据进行生存分布。这是一个非参数统计,它允许我们估计生存函数,因此不基于潜在的概率分布。 Kaplan-Meier 估计值基于治疗后存活一定时间的患者总数中的患者数量(每个患者作为一行数据)。 (这是事件)。
我们用公式表示 Kaplan-Meier函数:
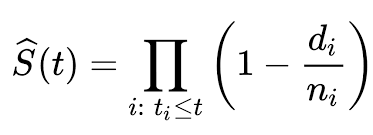
这里S(t)表示生命比t长的概率ti (至少发生了一个事件), di表示在时间ti发生的事件(例如死亡)的数量, ni表示存活到时间ti
例子:
我们将使用 Survival 包进行分析。使用预加载在生存包中的Lung数据集,该数据集包含来自 North Central 癌症治疗组的 228 名晚期肺癌患者的数据,基于 10 个特征。数据集包含缺失值,因此假设在构建模型之前在您身边完成缺失值处理。
R
# Installing package
install.packages("survival")
# Loading package
library(survival)
# Dataset information
?lung
# Fitting the survival model
Survival_Function = survfit(Surv(lung$time,
lung$status == 2)~1)
Survival_Function
# Plotting the function
plot(Survival_Function)R
# Installing package
install.packages("survival")
# Loading package
library(survival)
# Dataset information
?lung
# Fitting the Cox model
Cox_mod <- coxph(Surv(lung$time,
lung$status == 2)~., data = lung)
# Summarizing the model
summary(Cox_mod)
# Fitting survfit()
Cox <- survfit(Cox_mod)
# Plotting the function
plot(Cox)在这里,我们对“时间”和“状态”感兴趣,因为它们在分析中起着重要作用。时间代表患者的生存时间。由于患者存活,我们将他们的状态视为死亡或未死亡(审查)。
Surv()函数将两次和状态作为输入,并创建一个对象作为survfir()函数的输入。我们在survfit()函数中传递 ~1 以确保我们告诉函数根据生存对象拟合模型并产生中断。
survfit()创建生存曲线并打印值的数量、事件的数量(患有癌症的人)、中位时间和 95% 置信区间。该图提供以下输出:
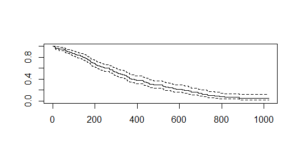
这里,x 轴指定“天数”,y 轴指定“生存概率”。虚线是上置信区间和下置信区间。
我们还有显示预期误差幅度的置信区间,即在存活 200 天的天数内,置信区间上限达到 0.76 或 76%,然后下降到0.60 或 60% 。
Cox 比例风险模型
它是一种回归模型,用于衡量瞬时死亡风险,比 Kaplan-Meier 估计量更难说明。它由风险函数h(t)组成,它描述了在特定时间t之前事件或风险h (例如生存)的概率。危险函数考虑协变量(回归中的自变量)来比较患者组的生存率。
它不假设潜在的概率分布,但它假设我们比较的患者组的危害随着时间的推移是恒定的,因此它被称为“比例风险模型”。
例子:
我们将使用 Survival 包进行分析。使用预加载在生存包中的Lung数据集,该数据集包含来自 North Central 癌症治疗组的 228 名晚期肺癌患者的数据,基于 10 个特征。数据集包含缺失值,因此假设在构建模型之前在您身边完成缺失值处理。我们将使用 cox 比例风险函数coxph()来构建模型。
R
# Installing package
install.packages("survival")
# Loading package
library(survival)
# Dataset information
?lung
# Fitting the Cox model
Cox_mod <- coxph(Surv(lung$time,
lung$status == 2)~., data = lung)
# Summarizing the model
summary(Cox_mod)
# Fitting survfit()
Cox <- survfit(Cox_mod)
# Plotting the function
plot(Cox)
在这里,我们对“时间”和“状态”感兴趣,因为它们在分析中起着重要作用。时间代表患者的生存时间。由于患者存活,我们将他们的状态视为死亡或未死亡(审查)。
Surv()函数将两次和状态作为输入,并创建一个对象作为survfir()函数的输入。我们在survfit()函数中传递 ~1 以确保我们告诉函数根据生存对象拟合模型并产生中断。
Cox_mod 输出类似于回归模型。有一些重要的特征,如年龄、性别、ph.ecog 和 wt。损失。该图提供以下输出:
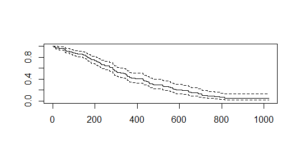
这里,x 轴指定“天数”,y 轴指定“生存概率”。虚线是上置信区间和下置信区间。与 Kaplan-Meier 图相比,Cox 图的初始值较高,较高的值较低,因为 Cox 图中有更多变量。
我们还有显示预期误差幅度的置信区间,即在存活 200 天的天数内,置信区间上限达到0.82 或 82% ,然后下降到0.70 或 70%。
注意: Cox 模型比 Kaplan-Meier 提供更好的结果,因为它的数据和特征最不稳定。 Cox 模型对于较低的值也较高,反之亦然,即随着时间的增加急剧下降。